
UNIST(총장 이용훈)는 남덕우 생명과학과 교수팀이 단일세포 시퀀싱 데이터 통합분석으로 폐암이나 각종 감염병의 질병 관련 유전자를 효과적으로 선별해낼 수 있음을 입증했다고 13일 밝혔다.
단일세포 시퀀싱(scRNA-seq)은 개별 세포 단위에서 유전자 발현을 분석하는 기술이다. 각종 질병, 발생, 분화 등 생물학적 과정을 분석하는 데 널리 활용된다. 세포 유형별 순일한 유전자 발현 변화를 측정해 질병 발생 기작을 보다 정확하게 분석할 수 있다.
하지만 단일세포 데이터의 높은 잡음과 결측률, 데이터 간 측정값 차이(배치효과)로 인해 실제 질병 유전자 분석에 얼마나 효과적인지는 제대로 확인되지 않았다.
남 교수팀은 다양한 시뮬레이션과 단일세포 데이터 분석 등 46개 통합분석 방법을 비교하고, 1기 폐암환자의 단일세포(상피세포) 데이터를 확보해 세포유형별로 통합 분석했다.
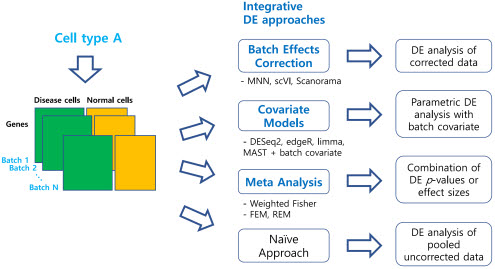
그 결과 기존 폐암 관련 유전자로 보고된 90여개 유전자가 통계적으로 높은 순위에 있다는 점을 확인했다. 이를 통해 아직 밝혀지지 않은 폐암 유전자도 높은 순위로 선별할 수 있다는 것과 이러한 단일세포 통합분석이 폐암유전자 발굴에 훨씬 효과적이라는 것을 입증했다.
남 교수팀은 대규모 COVID-19 환자 샘플에서 추출한 단핵구 세포 10만개 데이터를 추가 분석하고, 단일세포 데이터 통합분석이 다른 유형의 질병에서도 유전자를 효과적으로 선별할 수 있다는 것을 확인했다.
남덕우 교수는 “공개된 단일세포 데이터를 대규모로 통합분석해 새로운 질병유전자와 작용 경로를 찾아냈다”며 “암, COVID-19 등 다양한 질병의 기작 연구에서 단일세포 데이터 분석을 세포 수준을 넘어 유전자 수준까지 적극 활용할 필요가 있다”고 말했다.
이번 연구결과는 '네이처 커뮤니케이션즈' 3월 21일자에 실렸다.
울산=임동식기자 dslim@etnews.com









