광주과학기술원(GIST)은 이현주 인공지능(AI)대학원 교수팀이 단일세포 리보핵산(RNA) 시퀀싱 기술의 근본적인 한계를 극복할 수 있는 자기 지도 학습 방법론을 개발했다고 25일 밝혔다.
연구팀은 이를 적용해 동일한 세포 종류라도 당뇨병의 정도에 따라 구분되는 세부적인 특징까지도 발견할 수 있었다. 15개의 단일세포 RNA 데이터셋에서 실시한 세포 종류 분류 테스트 중 12개 데이터셋에서 가장 높은 F1 점수를 보였다. F1 점수는 정밀도와 재현율의 균형을 평가하는 점수다.
여러 세포의 RNA가 섞인 유전자 발현량을 측정할 수 있는 다세포 RNA 시퀀싱 기술에 반해 단일세포 RNA 시퀀싱은 단일세포만을 대상으로 하기 때문에 측정 정확도가 떨어진다. 단일세포 RNA 데이터는 보통 3만개가 넘는 유전자 중에서 2000~3000개 유전자의 발현량만을 얻는 경우가 많다. 즉, 전체 유전자 가운데 10%만이 측정 가능한 높은 해상도를 가지고 나머지 90% 정보는 낮은 해상도로 인해 측정이 불가하다.
기존 연구는 주로 여러 세포에서 공통으로 발현되는 약 10%의 유전자만을 사용하여 세포 유형을 예측하고 분석해 왔다. 하지만 특정 세포 종류에서만 발현되는 유전자가 오히려 해당 세포를 더 자세히 설명하는 경우가 많다. 현재 사용하는 단일세포 RNA 시퀀싱 기술은 약 90%의 유전자 정보를 사용하지 못하는 근본적인 문제점이 있다.
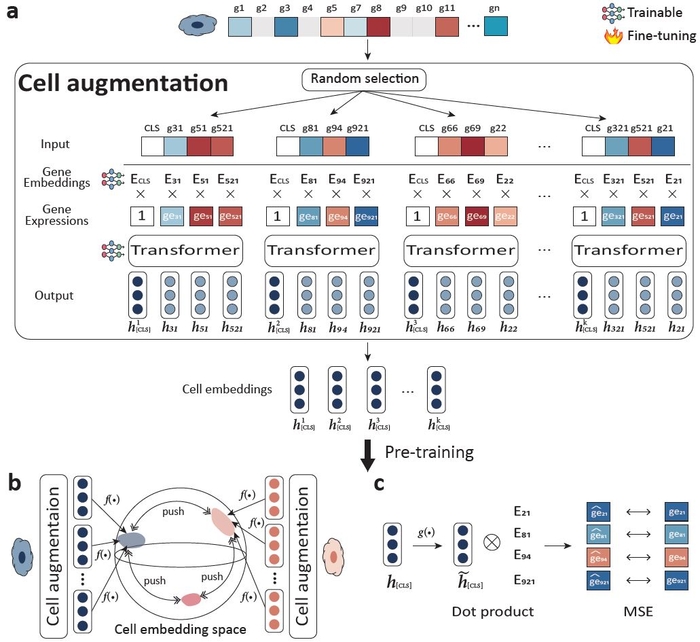
연구팀은 단일세포 RNA 시퀀싱 데이터에 적합한 자기 지도 학습 방법론 가운데 하나인 대조 학습 방법론을 활용해 단 5% 미만의 유전자 정보만을 가지고도 각 단일세포의 보편적인 특징부터 세부적인 특징까지도 파악할 수 있는 기술인 '에스씨로버스터(scRobust)'를 개발했다. 이전 방법론에서 활용되지 않았던 90%의 유전자 정보까지 사용할 수 있게 돼 세포 종류에 대한 예측 성능이 향상되었을 뿐만 아니라 동일한 세포 내에서도 더욱 정밀한 분석이 가능해졌다.

이 기술은 하나의 세포로부터 다양한 유전자 조합을 만들어 여러 개의 세포 표현 벡터를 생성할 수 있는 방법론을 기반으로 단일세포 RNA 시퀀싱 데이터에 적합한 데이터 증강을 하는 것이다. 대조 학습으로 AI 모델을 학습시키면 서로 다른 유전자 조합으로 생성된 세포 표현 벡터라도 같은 세포에서 나온 것인지, 다른 세포에서 나온 것인지 구분할 수 있다. 이 과정을 통해 다양한 유전자 조합으로 만든 세포 표현 벡터들이 하나의 통일된 세포 표현 벡터로 수렴하게 된다. 결과적으로 소수의 유전자만 사용하더라도 모든 유전자를 활용한 것과 유사한 세포 표현 벡터를 얻을 수 있어 전체 유전자를 사용하는 효과를 기대할 수 있다.
이현주 교수는 “이번 연구에서 개발된 알고리즘은 AI 모델이 유전자 일부만 학습하는 것이 아니라 모든 유전자에 대해 학습하는 것이 가능하다”면서 “이를 통해 그동안 소수의 세포에서만 발현되는 유전자와 같은 세포 유형에서 발생하는 미세한 특징들까지도 비교, 분석할 수 있게 되었다”고 말했다.
또한 “다양한 세포 종류의 마커 유전자뿐만 아니라 약물 저항성과 관련된 마커 유전자까지 추출할 수 있어 향후 단일세포 분석의 패러다임을 바꿀 수 있을 것으로 기대한다”고 덧붙였다.
광주=김한식 기자 hskim@etnews.com









