국내 연구진이 유전체 분석을 더욱 빠르게 할 컴퓨팅 시스템 기술을 개발했다.
한국전자통신연구원(ETRI) 유전체 분석에 특화된 메모리 중심 컴퓨팅 시스템을 개발했다고 23일 밝혔다. 기존 대비 28% 성능 향상을 이뤘다.
유전체 분석을 활용하면 개인별 질병 위험도, 영양과 운동 상호작용 등을 알 수 있다. 하지만 아직 서비스를 대중화하기에는 단가가 비싸고 데이터 양도 커서 많은 비용이 필요하다.
ETRI는 유전체를 분석하는 차세대 염기서열분석(NGS)에 특화된 메모리 중심 컴퓨팅 하드웨어(HW) 및 소프트웨어(SW) 기술을 개발했다.
그간 유전체 분석은 주로 메모리를 제한적으로 사용하되 연산을 많이 하는 프로세서 중심 컴퓨팅 기술을 주로 쓴다. 유전체 분석처럼 대용량 데이터를 처리할 때는 병목 현상이 일어나 데이터 처리에 시간과 노력이 많이 든다.
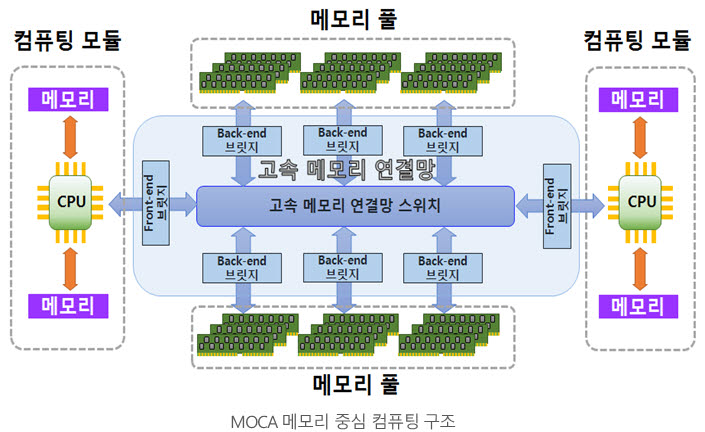
ETRI 메모리 중심 컴퓨팅 기술은 대규모 메모리를 활용해 병목 현상을 극복했다. 자체 개발한 'MOCA'라는 HW장치로 대규모 메모리를 시스템에 장착할 수 있게 했다. 데이터 처리 중간 과정에서 하드디스크나 SSD 등을 활용할 필요가 없게 만든 것이 핵심이다.
또 연구진은 오랜 시간이 걸리는 염기 서열 정렬 단계를 대규모 메모리를 활용해 2배 이상 빠르게 처리할 수 있는 SW도 개발해 분석 효율을 높였다.
GC녹십자지놈과 협력해 기술 성능도 검증했다. 기존 시스템에 연구진이 개발한 HW를 적용하면 전체 분석 성능을 16% 높일 수 있고 HW와 SW를 동시에 적용하면 28%까지 성능을 향상할 수 있음을 보였다.
이 기술은 암 발병률, 태아 장애 유무 등을 미리 알아보거나 전염병 변이 파악, 치료제 개발 등을 하기 위한 시스템에 적용할 수 있다.
김강호 ETRI 데이터중심컴퓨팅시스템연구실장은 “이 기술이 국내 제약 분석 시장 및 산업에 새로운 촉진제가 되어 다양한 바이오 응용 시장 활성화 및 고용 효과가 매우 클 것으로 예상된다”고 말했다.
향후 연구진은 내년부터 2단계로 시작되는 '메모리 중심 차세대 컴퓨팅 시스템 구조 연구'과제에서 시스템을 고도화하고 의료기관을 확대해 유전체 분석 정확도를 더욱 높이는 한편, 암이나 당뇨병 등 다른 질병에도 적용 범위를 확대할 계획이다.
연구진은 이번 성과와 관련해 국제전기전자공학회(IEEE)가 발간하는 세계적 학술지를 비롯해 논문 26편, 국내외 특허 12건 출원, 기술이전 2건을 진행한 바 있다.
김영준기자 kyj85@etnews.com









